Lehman, Maurice J. Bessman, Ernest S. Simms, and Arthur
KornbergEnzymatic Synthesis of Deoxyribonucleic Acid: I. PREPARATION OF SUBSTRATES AND PARTIAL PURIFICATION OF AN ENZYME FROM ESCHERICHIA COLI // J. Biol. Chem. 1958 233: 163-170.
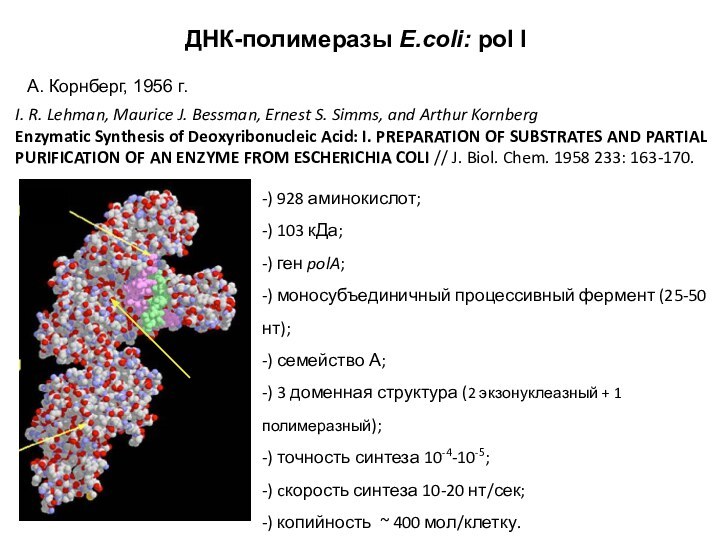
-) 928 аминокислот;
-) 103 кДа;
-) ген polA;
-) моносубъединичный процессивный фермент (25-50 нт);
-) семейство А;
-) 3 доменная структура (2 экзонуклеазный + 1 полимеразный);
-) точность синтеза 10-4-10-5;
-) cкорость синтеза 10-20 нт/сек;
-) копийность ~ 400 мол/клетку.